Modelo computacional arroja posibles compuestos que inhiben la covid-19
Se trata de un artículo de investigación que señala ciertas drogas como potentes candidatos antivirales contra el SARS-CoV-2. Los resultados fueron arrojados por un modelo de relaciones cuantitativas (QSAR) y los compuestos con menor concentración inhibitoria son la niclosamida, salinomicina, digitoxina y digoxina.
En diciembre de 2019 se detectó el primer caso de la enfermedad por covid-19 en Wuhan, China. Hasta la fecha, la pandemia por coronavirus ha sido causante de más de tres millones de muertes, el colapso de los sistemas de salud y el estancamiento de la economía. Por estos y otros motivos, el virus SARS-CoV-2 ha sido objeto de estudio de múltiples investigaciones alrededor del mundo que han resultado en grandes hallazgos científicos.
Los diferentes grupos de investigación de la Universidad del Norte se han vinculado a esta causa global, entre ellos, el Grupo de Investigaciones en Química y Biología, coordinado por Edgar Márquez Brazón, licenciado en Química con postdoctorado en Biotecnología Farmacéutica de la Universidad Católica de Portugal.
Desde marzo del año pasado, el docente del departamento de Química y Biología se propuso investigar formas efectivas de combatir el coronavirus, un trabajo que desarrolló en alianza con José Ramón Mora, docente del departamento de Ingeniería Química de la Universidad San Francisco de Quito e investigador del Instituto de Investigaciones Atmosféricas, y Sebastián Cuesta, asistente de investigación del Instituto de Simulación Computacional de Quito.
Los investigadores estudiaron varios compuestos que presentan actividad biológica in vitro contra la covid. A partir de cálculos computacionales, determinaron las características fisicoquímicas que eran necesarias para que estos compuestos inhibieran las proteínas fundamentales en la proliferación del virus SARS-CoV-2, creando un modelo de Relaciones Cuantitativas Estructura-Actividad (QSAR). Teniendo en cuenta los datos arrojados por el modelo matemático, buscaron en la base de datos DRUGSBANK, que contiene los compuestos aprobados por de la Administración de Alimentos y Medicamentos (FDA), aquellos que por sus características pueden ser candidatos potenciales anti COVID-19.
“De la base de datos mundial de la FDA extrajimos los compuestos con características fisicoquímicas causantes de inhibir las proteínas fundamentales del virus. Al encontrar estas características, buscamos entre las 6.664 drogas aprobadas por la FDA aquellas que tienen la capacidad de inhibir por lo menos una de las proteínas”, señala el investigador venezolano Márquez Brazón.
Añade que “el modelo computacional arrojó un total de 664 moléculas, y discriminamos entre aquellas que tienen una menor concentración inhibitoria hasta llegar a 54 compuestos que son potentes candidatos antivirales contra el SARS-CoV-2. Entre estos, encontramos antibióticos y antivirales. Los compuestos con los porcentajes más altos son la niclosamida, salinomicina, digitoxina y digoxina”.
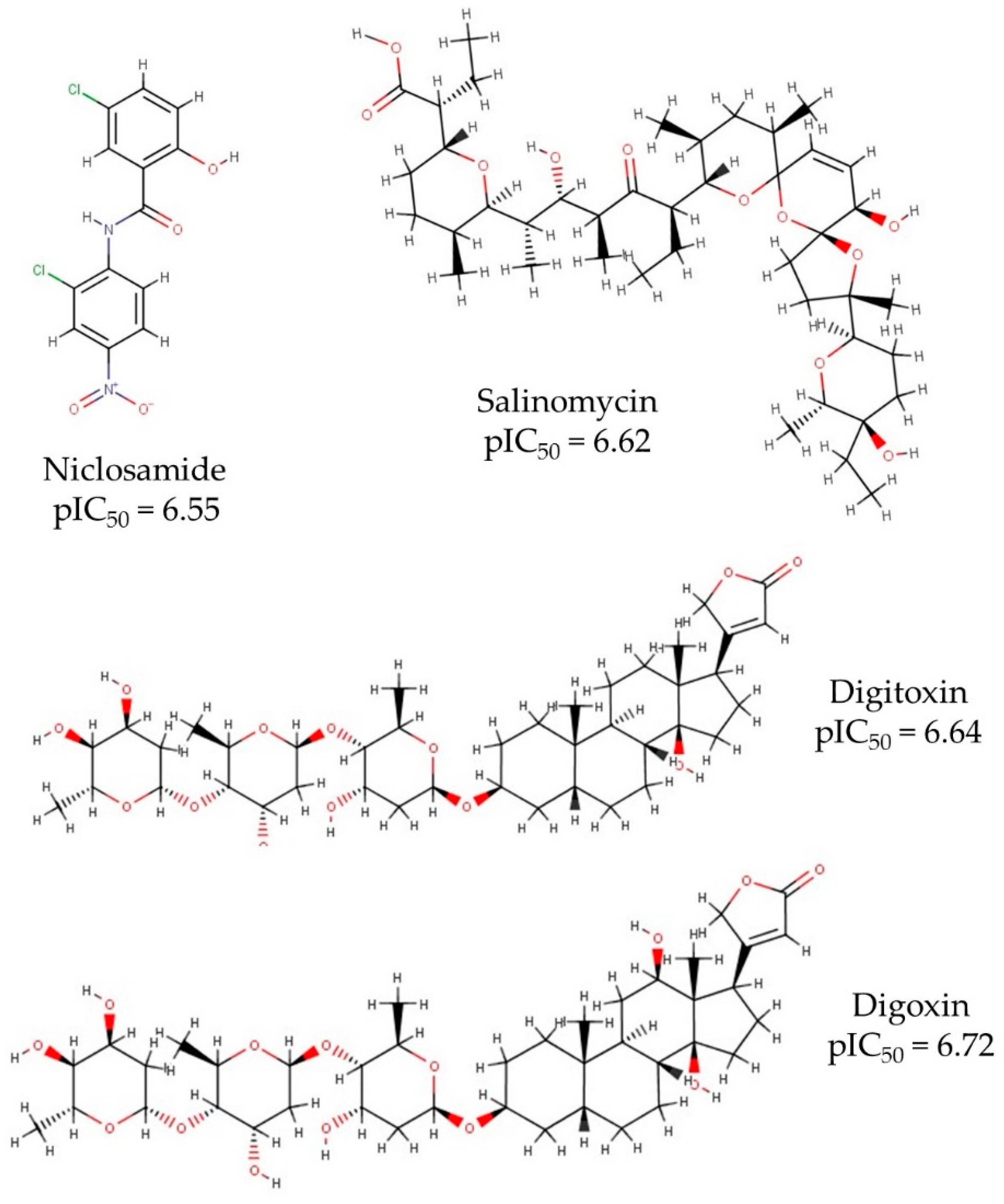 |
Los cuatro candidatos antivirales más potentes, basados en valores experimentales.
El artículo fue publicado el 19 de febrero de este año, y hace aproximadamente dos meses una investigación divulgada por la revista Lancet demostró que la niclosamida (el compuesto con la menor concentración inhibitoria que arrojó el modelo matemático) es un antihelmíntico antiviral efectivo contra el virus y ya se encuentra en la fase III de los tratamientos empleados para controlar la covid. Lo que implica que este modelo es capaz de predecir aquellas drogas aprobadas por la FDA que pueden ser probadas contra la covid.
“Gracias a este tipo de trabajos, los investigadores hemos reducido entre 20 y 25 años para la aprobación de compuestos que son útiles en el tratamiento de ciertas enfermedades, pues predecimos el potencial de drogas aprobadas por la FNA que los científicos pueden probar mediante ensayos in vitro o in vivo. De esta manera, las drogas se ponen en fase II y III rápidamente. Este tipo de técnicas son usadas por las empresas farmacéuticas para el diseño de nuevos compuestos y la recomposición de drogas”, acota el docente de la División de Ciencias Básicas.
Los resultados muestran que, a través de la química computacional, se pueden generar datos muy confiables y proponer estructuras que puedan ser inhibidoras de un virus. Además, el grupo de investigación en Química y Biología de Uninorte ha venido trabajando con metodologías computacionales desde hace años, lo que les ha permitido predecir compuestos con propiedades anticancerígenas, antituberculosis, entre otras enfermedades.
Puede consultar el artículo completo aquí.
Por María Fernanda Salgado.
Más noticias
Abr 26, 2024
Histórico

Abr 26, 2024
Histórico

Abr 26, 2024
Histórico

 español
español inglés
inglés francés
francés alemán
alemán